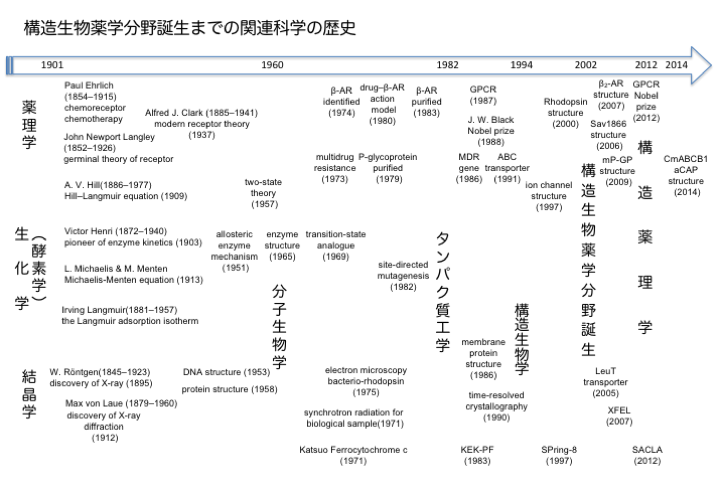
分子生物学から構造生物学そして構造薬理学へ
1940年代終わりに生物の機能を分子構造から理解することを目的として、X線結晶学によるタンパク質とDNAの分子構造決定から始まった分子生物学は、 その後、遺伝子からアプローチする科学として分子遺伝学を指すようになり、分子構造から生物を考える科学は、1990年代から「構造生物学」と呼ばれるようになる1)。 タンパク質のX線結晶解析は、当初から材料が豊富に入手可能な多くの酵素に応用され、1980年代までには触媒化学反応が詳細に理解された。 その後、遺伝子工学の発展によって存在量の少ないタンパク質の大量調製が可能となる一方、 発現領域の設計やアミノ酸変異導入も自在に行うことが可能となり調製が困難であったタンパク質も結晶化が可能になった。 この結果、情報伝達2), 3)、遺伝子転写制御におけるDNA認識4)、免疫応答の分子認識5)など、 生物学的に重要な機能を担う分子の機能が分子構造に基づいて解明されるようになり、X線結晶学の中心は化学から生物学へと移っていった。 またちょうどこの頃、酵素反応の詳しい理解が進んだことが、酵素を標的とする阻害剤の設計を分子構造に基づいて行う、 いわゆるStructure-based Drug designを実現させた6), 7, 8)。そして、不治の病であったHIVの治療薬や抗インフルエンザ薬が誕生した。 もはや構造生物学は創薬に欠かせない研究手法となっている。
さらに結晶化技術の革新により、20世紀の終わりには受容体やトランスポーターなど膜に埋め込まれた膜タンパク質の立体構造研究が盛んになり、 今日では、生理学的な機能やリガンドに対する薬理学的な作用を立体構造に基づいて、議論することが可能になってきた。 1985年に膜タンパク質のX線結晶解析が達成されて以来9)、天然から豊富に得られる分子を標的としてロドプシン10), 11) やカルシウムポンプ12) などの立体構造解析が長い期間をかけて達成された。一方、ヒトと機能が類似した膜タンパク質を細菌から探して、 機能を保持したまま結晶化に適した領域だけを標的とすることで、カリウムイオンチャネルの立体構造解析が達成された13)。 さらに、特筆すべき技術革新が行われた。すなわち、立体構造を認識する抗体による結晶格子中の立体構造の安定化技術14)、 そして、lipidic cubic phase法による脂質中を模した環境下での結晶化技術15)、 さらに、蛍光ゲルろ過法を用いた結晶化に適した遺伝子のスクリーニング技術である16)。 これらの技術により、膜を介するトランスポーター17)の立体構造解析が短期間に行われるようになった。
20世紀初頭にJ. N. Langley18)らが提唱した受容体説は、薬理学のセントラルドグマとなっている。 もっとも、当初は薬物に対する生体の応答から考案された仮説であり、徐々に認められてはきたが、受容体の実態は50年を経ても不明のままだった19)。 しかし、70年近い時を経てアイソトープラベルされたアンタゴニストを用いた実験で細胞膜に実際に分子として存在することが確認された20), 21)。 そして、アドレナリン受容体がアンタゴニストを用いた親和性クロマトグラフィーを用いて単離精製され22) 、 明らかになったアミノ酸部分配列をもとに遺伝子がクローニングされた23) 。このとき、ロドプシンとの類似性に気づいたR. J. Lefkowitzによって、 Gタンパク質共役型受容体(GPCR)と命名されるに至っている。そしてついに、2007年、B. K. Kobilkaらによって結晶解析が達成された24)。 さらに、三量体Gタンパク質との複合体の構造も明らかとなり、薬理学的解析から示唆されていた分子応答と受容体の構造変化の関係が分子構造から説明された25)。 さらに、NMRを用いて構造の動きと機能の関係も解明されつつある26) , 27)。
2012年にはGPCRの研究に対してノーベル化学賞が授与されたことは記憶に新しい。 今やGPCRでは、受容体の分子構造を基に、アゴニストとアンタゴニストの違いはもちろん、 完全アゴニストと部分アゴニストあるいは逆アゴニストの作用メカニズムの違いも理解できるようになってきた。 構造生物学の波は薬理学にも及んだ。構造薬理学の誕生である28), 29)。
生命現象のメカニズムの理解は分子構造がわかるだけでは不十分であり、分子構造研究と機能研究の統合的な研究の推進が重要になるであろう。 生物機能の研究者が自ら構造を解析することがもっと広がれば、さらに新たな発見の可能性が高まる。 なぜなら、機能を念頭において立体構造を眺めると、天啓が得られることを構造生物学研究者の多くが経験しているからである。 反対に機能を理解せず構造を眺めても、単なる丸や四角にしか見えない。データバンク(Protein Data Bank)に登録されているGPCRの立体構造は、100を超えている。 もはや立体構造研究は特殊な分子の例ではない時代を迎えている。立体構造情報の有効活用法をもっと真剣に考えるべきときに来ているという思いを共有し、 多様な専門家と協力して今後の薬学研究の展開を図りたい。
References
1) Campbell, I. D., Nat Rev Mol Cell Biol, 3, 377-381 (2002).
2) Wall, M. A., Coleman, D. E., Lee, E., Iniguez-Lluhi, J. A., Posner, B. A., Gilman, A. G., Sprang, S. R., Cell, 83, 1047-1058 (1995).
3) Lambright, D. G., Sondek, J., Bohm, A., Skiba, N. P., Hamm, H. E., Sigler, P. B., Nature, 379, 311-319 (1996).
4) Ferre-D’Amare, A. R., Prendergast, G. C., Ziff, E. B., Burley, S. K., Nature, 363, 38-45 (1993).
5) Madden, D. R., Garboczi, D. N., Wiley, D. C., Cell, 75, 693-708 (1993).
6) Wlodawer, A., Miller, M., Jaskolski, M., Sathyanarayana, B. K., Baldwin, E., Weber, I. T., Selk, L. M., Clawson, L., Schneider, J., Kent, S. B., Science, 245, 616-621 (1989).
7) Roberts, N. A., Martin, J. A., Kinchington, D., Broadhurst, A. V., Craig, J. C., Duncan, I. B., Galpin, S. A., Handa, B. K., Kay, J., Krohn, A., et al., Science, 248, 358-361 (1990).
8) von Itzstein, M., Wu, W. Y., Kok, G. B., Pegg, M. S., Dyason, J. C., Jin, B., Van Phan, T., Smythe, M. L., White, H. F., Oliver, S. W., et al., Nature, 363, 418-423 (1993).
9) Deisenhofer, J., Epp, O., Miki, K., Huber, R., Michel, H., Nature, 318, 618-624 (1985).
10) Okada, T., Le Trong, I., Fox, B. A., Behnke, C. A., Stenkamp, R. E., Palczewski, K., J Struct Biol, 130, 73-80 (2000).
11) Palczewski, K., Kumasaka, T., Hori, T., Behnke, C. A., Motoshima, H., Fox, B. A., Le Trong, I., Teller, D. C., Okada, T., Stenkamp, R. E., Yamamoto, M., Miyano, M., Science, 289, 739-745 (2000).
12) Toyoshima, C., Nakasako, M., Nomura, H., Ogawa, H., Nature, 405, 647-655 (2000).
13) Doyle, D. A., Morais Cabral, J., Pfuetzner, R. A., Kuo, A., Gulbis, J. M., Cohen, S. L., Chait, B. T., MacKinnon, R., Science, 280, 69-77 (1998).
14) Iwata, S., Ostermeier, C., Ludwig, B., Michel, H., Nature, 376, 660-669 (1995).
15) Landau, E. M., Rosenbusch, J. P., Proc Natl Acad Sci U S A, 93, 14532-14535 (1996).
16) Kawate, T., Gouaux, E., Structure, 14, 673-681 (2006).
17) Yamashita, A., Singh, S. K., Kawate, T., Jin, Y., Gouaux, E., Nature, 437, 215-223 (2005).
18) Langley, J. N., J. Physiol (London), 33, 374 – 413 (1905).
19) Maehle, A. H., Endeavour, 33, 135-140 (2009).
20) Lefkowitz, R. J., Roth, J., Pricer, W., Pastan, I., Proc Natl Acad Sci U S A, 65, 745-752 (1970).
21) Alexander, R. W., Williams, L. T., Lefkowitz, R. J., Proc Natl Acad Sci U S A, 72, 1564-1568 (1975).
22) Caron, M. G., Srinivasan, Y., Pitha, J., Kociolek, K., Lefkowitz, R. J., J. Biol. Chem., 254, 2923-2927 (1979).
23) Dixon, R. A., Kobilka, B. K., Strader, D. J., Benovic, J. L., Dohlman, H. G., Frielle, T., Bolanowski, M. A., Bennett, C. D., Rands, E., Diehl, R. E., Mumford, R. A., Slater, E. E., Sigal, I. S., Caron, M. G., Lefkowitz, R. J., Strader, C. D., Nature, 321, 75-79 (1986).
24) Cherezov, V., Rosenbaum, D. M., Hanson, M. A., Rasmussen, S. G., Thian, F. S., Kobilka, T. S., Choi, H. J., Kuhn, P., Weis, W. I., Kobilka, B. K., Stevens, R. C., Science, 318, 1258-1265 (2007).
25) Rasmussen, S. G., DeVree, B. T., Zou, Y., Kruse, A. C., Chung, K. Y., Kobilka, T. S., Thian, F. S., Chae, P. S., Pardon, E., Calinski, D., Mathiesen, J. M., Shah, S. T., Lyons, J. A., Caffrey, M., Gellman, S. H., Steyaert, J., Skiniotis, G., Weis, W. I., Sunahara, R. K., Kobilka, B. K., Nature, 477, 549-555 (2011).
26) Yoshiura, C., Kofuku, Y., Ueda, T., Mase, Y., Yokogawa, M., Osawa, M., Terashima, Y., Matsushima, K., Shimada, I., J Am Chem Soc, 132, 6768-6777 (2010).
27) Manglik, A., Kim, T. H., Masureel, M., Altenbach, C., Yang, Z., Hilger, D., Lerch, M. T., Kobilka, T. S., Thian, F. S., Hubbell, W. L., Prosser, R. S., Kobilka, B. K., Cell, 161, 1101-1111 (2015).
28) Piscitelli, C. L., Kean, J., de Graaf, C., Deupi, X., Mol. Pharm., (2015).
29) Kristensen, A. S., Andersen, J., Jorgensen, T. N., Sorensen, L., Eriksen, J., Loland, C. J., Stromgaard, K., Gether, U., Pharm. Rev, 63, 585-640 (2011).